围绕一个蛋白质分子机器,机制研究能够做到多深入?近日,清华大学娄智勇教授/饶子和院士、中国医学科学院北京协和医院朱兰教授团队与广州国家实验室、南开大学等单位合作,揭示了冠状病毒转录复制过程中RNA模板循环和RNA加帽的共发生机制,重构了病毒领域对RNA病毒转录复制过程的认知。该工作于2025年10月22日在Cell在线发表。这是团队在冠状病毒转录复制机制的研究中,自2020以来在Science、Cell等期刊上连续发表10项成果后的又一重要工作。
RNA病毒是一种极其简单但高效的生命形式。在生命周期中,RNA病毒利用有限的基因组,编码几个到十几个蛋白质,即可完成高效扩增,这要求病毒基因组必须能够高效的循环使用。同时,转录形成的新生RNA 5’端带有三磷酸基团,一旦释放到宿主细胞中,会迅速激活宿主细胞的先天免疫反应,因此必须立即对其5’端的三磷酸基团进行加工,形成“帽”(Cap)结构,从而逃逸宿主天然免疫的攻击。虽然基因组的高效重复使用、RNA加帽是RNA病毒生命过程中最核心、最基础的生物学事件,但自从100多年前发现第一种RNA病毒至今,这两个过程协同发生的机制一直是病毒学领域的关键未解之谜。
围绕冠状病毒转录复制过程,研究团队在过去5年中先后阐明了“核心转录复制复合体”(C-RTC)[1]、“延伸复合体”(E-RTC)[2]、“加帽中间态复合体”[3, 4]和“Cap0加帽复合体”[5]的工作机制。在这些工作的基础上,研究团队设计了多种全新核酸骨架,成功捕捉到转录复制复合体“加帽起始前状态”(Pre-Capping Initiation, Pre-CI)和“加帽起始后状态”(Post-Capping Initiation, Post-CI)两个超级复合体,阐明了RNA模板循环和RNA加帽起始过程协同发生的分子机制(图1)。
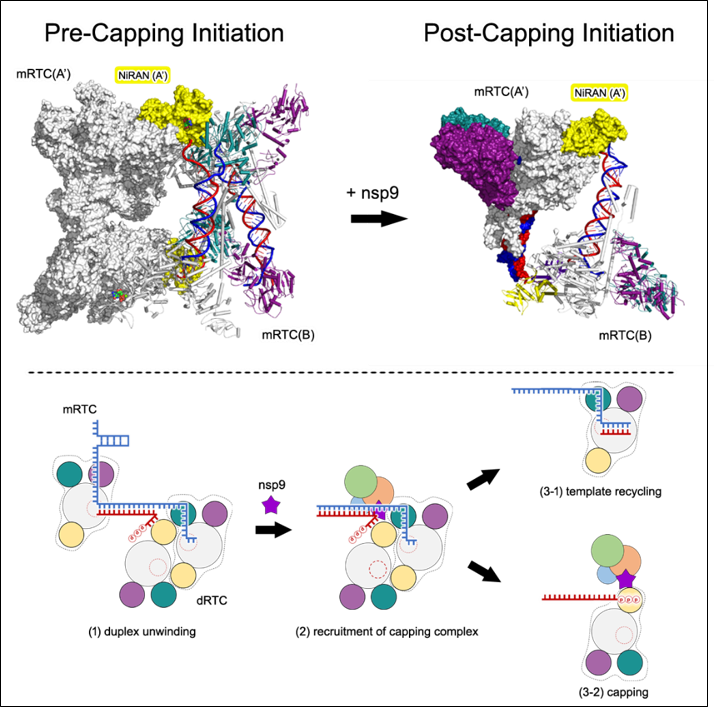

图1. 冠状病毒“模板循环”和“RNA加帽起始”共发生机制的模型
在该工作中,研究团队首先设计合成了具有39 bp配对双链的RNA骨架,利用高活性的新冠病毒解旋酶和其他转录复制酶,共同组装成“加帽起始前状态”复合体(图2)。该复合体包含四个延伸复合体(E-RTC),其中两个延伸复合体形成“背靠背”二聚体形态,再由两个二体以“头对头”的模式形成四聚体形态的超级复合体。在超级复合体中,由聚合酶nsp12催化位点延伸出来的“模板-产物”双链RNA,被处于“头对头”位置的延伸复合体中的解旋酶解开为单链,并进一步将模板RNA传递至“头对头”位置的延伸复合体中的聚合酶催化反应中心,从而实现了模板RNA的循环再利用。
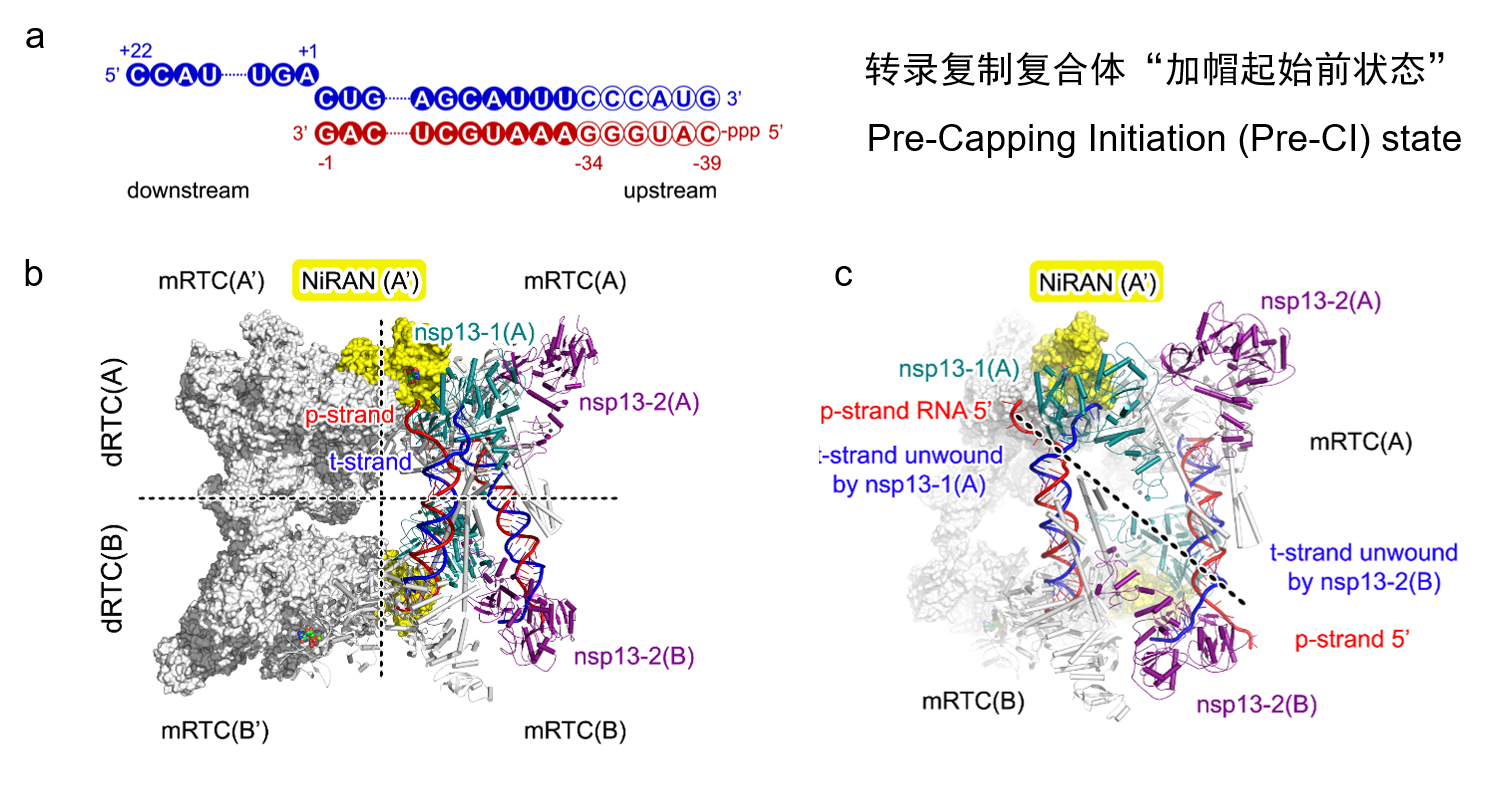
图2. (a)组装加帽起始前状态复合体所使用的RNA骨架。(b)加帽起始前状态复合体的四聚体全貌。(c)加帽起始前状态复合体中“模板-产物”双链RNA的解旋。
在“加帽起始前状态”复合体中,另一关键性发现是团队首次确定了冠状病毒解旋酶3’-5’的解旋极性,明确了解旋的关键结构单元,展现了由ATP水解驱动的双链RNA解旋动态过程(图3)。在冠状病毒研究领域,自从2000年第一次确定其解旋酶以来,一直认为其解旋极性是5’-3’,但由于和许多认知相冲突,一直备受争议。这项工作利用结构生物学手段,直观、明确的表明冠状病毒解旋酶的解旋方向,终结了对这一问题的争论。
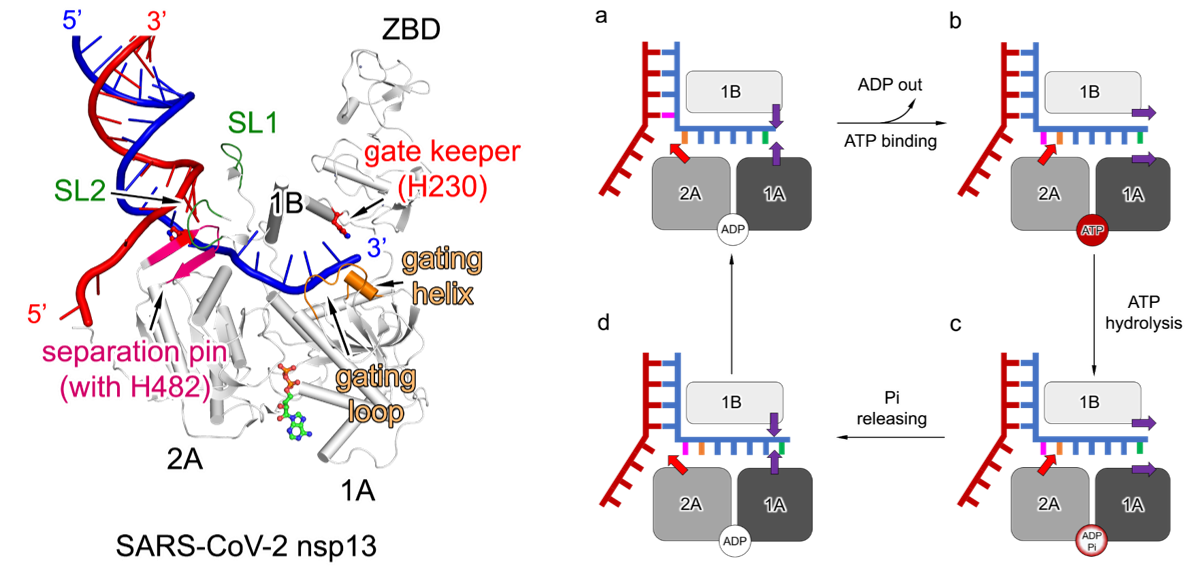
图3. 左图,新冠病毒解旋酶的关键结构单元。右图,ATP水解驱动的双链RNA解旋模式图。
随后,研究团队设计合成了具有非配对双链的RNA骨架,通过在体系中加入nsp9蛋白,捕捉到“加帽起始后状态”的复合体(图4)。加入nsp9蛋白后,复合体由加帽起始前状态的四聚体形态,转变为加帽起始后状态下的二聚体形态。最关键的一点是,RNA产物链5’端三磷酸集团,和nsp9蛋白N端氨基酸共定位于聚合酶NiRAN结构域的催化位点。在以往的研究中,一般认为冠状病毒的加帽过程由nsp13、nsp12 NiRAN结构域、nsp14、nsp16四个酶分子先后催化完成。2022年,研究团队发现了由nsp9 RNAylation介导的RNA加帽的全新机制[4],在该机制中,nsp12 NiRAN结构域可以不依赖nsp13,独立起始RNA加帽过程,但这两种加帽机制到底哪种更接近真实生命过程还存在争论。这项成果明确表明,新生RNA产物链5’端不会释放到溶液中、并由nsp13进行初步加工,而是直接进入nsp12 NiRAN结构域,通过nsp9 RNAylaion介导的方式起始RNA加帽过程,更新了对冠状病毒加帽过程的认知。也再一次以无可辩驳的结构生物学证据证明了团队发现的NiRAN催化、以GTP为底物的RNA加帽机制,终结了围绕NiRAN加帽机制的争论。
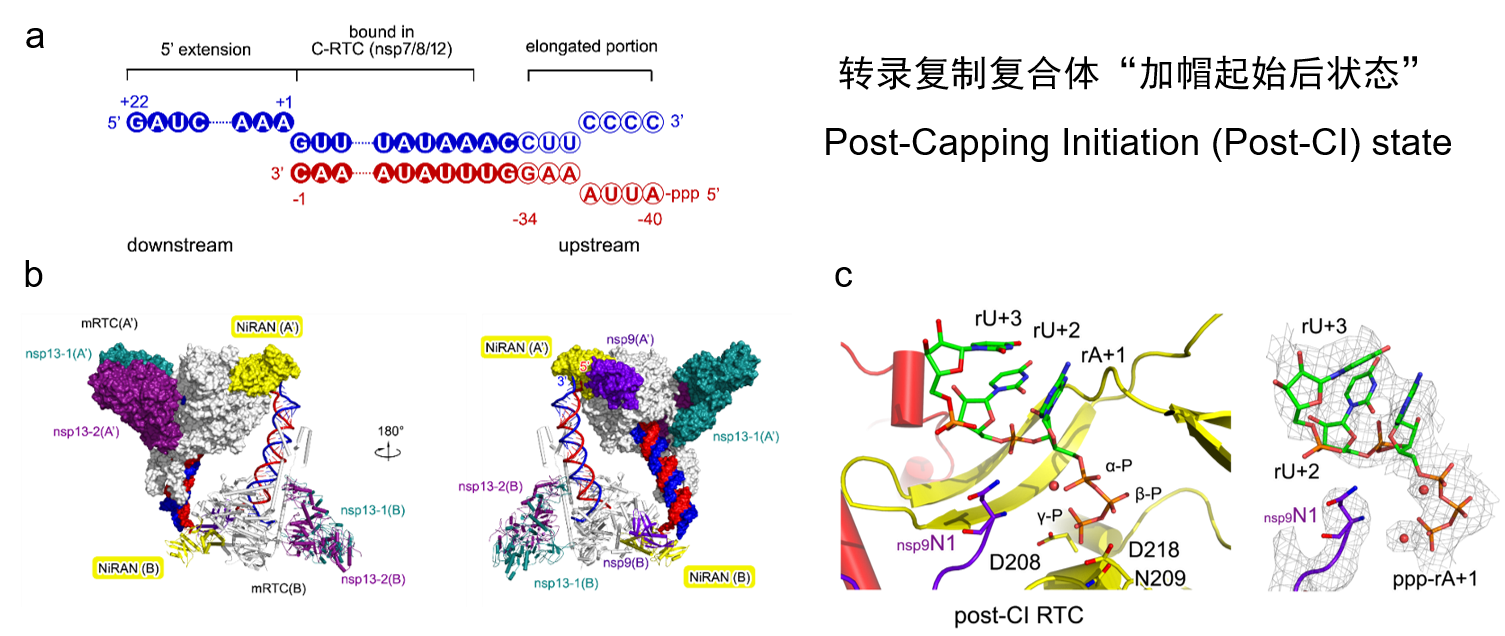
图4. (a)组装加帽起始后状态复合体所使用的RNA骨架。(b)加帽起始后状态复合体全貌。(c)加帽起始后状态复合体中RNA产物链5’三磷酸集团与nsp9蛋白共同定位与NiRAN结构域催化中心。
该成果重构了对病毒转录复制过程的认识体系,结束了对冠状病毒解旋酶极性、加帽机制等问题长达25年的争论。成果的获得得益于研究团队在冠状病毒转录复制领域中20余年的长期积累。自2020年以来,研究团队系统研究了新冠病毒转录复制过程,阐明了转录复制复合体多个状态三维结构,为认识病毒的生命过程、发展高效抗病毒药物提供了关键信息,还对新冠病毒对女性生殖道感染,以及新冠病毒灭活疫苗接种对母胎影响方面进行了深入研究,先后在Science(1)[1]、Cell上(6)[3-7]、Nature Communications(1)[2]和JACS(1)[8]、STTT(1)[9]和Clin Infect Dis(1)[10]上发表系列研究论文,是国际上新冠病毒转录复制研究中最为系统、引用最多的工作之一,成果入选 “中国科学十大进展”、“中国生命科学领域十大进展”、“中国2021年度医学重要进展”、“中华医学科技奖”等。
清华大学基础医学院娄智勇教授、饶子和院士和中国医学科学院北京协和医院的朱兰教授为共同通讯作者,清华大学基础医学院闫利明博士、南开大学药物化学生物学全国重点实验室(清华大学基础医学院已出站博士后)黄羽岑博士,南京医科大学生命科学学院(清华大学基础医学院已出站博士后)葛基博士,以及清华大学的刘奕潇、高山、谭丽萍、刘璐等为共同第一作者,研究工作得到南开大学、上海科技大学和清华大学冷冻电镜平台的支持,受到国家自然科学基金、广州国家实验室等的资助。
原文链接: https://www.cell.com/cell/abstract/S0092-8674(25)01090-6
参考文献
1. Gao Y., Yan L., Huang Y., Liu F., Zhao Y., Cao L., Wang T., Sun Q., Ming Z., Zhang L., Ge J., Zheng L., Zhang Y., Wang H., Zhu Y., Zhu C., Hu T., Hua T., Zhang B., Yang X., Li J., Yang H., Liu Z., Xu W., Guddat L.W., Wang Q., Lou Z., and Rao Z., Structure of the RNA-dependent RNA polymerase from COVID-19 virus. Science, 2020. 368(6492): p. 779-782.
2. Yan L., Zhang Y., Ge J., Zheng L., Gao Y., Wang T., Jia Z., Wang H., Huang Y., Li M., Wang Q., Rao Z., and Lou Z., Architecture of a SARS-CoV-2 mini replication and transcription complex. Nat Commun, 2020. 11(1): p. 5874.
3. Yan L., Ge J., Zheng L., Zhang Y., Gao Y., Wang T., Huang Y., Yang Y., Gao S., Li M., Liu Z., Wang H., Li Y., Chen Y., Guddat L.W., Wang Q., Rao Z., and Lou Z., Cryo-EM Structure of an Extended SARS-CoV-2 Replication and Transcription Complex Reveals an Intermediate State in Cap Synthesis. Cell, 2021. 184(1): p. 184-193 e10.
4. Yan L.M., Huang Y.C., Ge J., Liu Z.Y., Lu P.C., Huang B., Gao S., Wang J.B., Tan L.P., Ye S.H., Yu F.X., Lan W.Q., Xu S.Y., Zhou F., Shi L., Guddat L.W., Gao Y., Rao Z.H., and Lou Z.Y., A mechanism for SARS-CoV-2 RNA capping and its inhibition by nucleotide analog inhibitors. Cell, 2022. 185(23): p. 4347-+.
5. Yan L.M., Yang Y.X., Li M.Y., Zhang Y., Zheng L.T., Ge J., Huang Y.C., Liu Z.Y., Wang T., Gao S., Zhang R., Huang Y.Y.Y., Guddat L.W., Gao Y., Rao Z.H., and Lou Z.Y., Coupling of N7-methyltransferase and 3′-5′ exoribonuclease with SARS-CoV-2 polymerase reveals mechanisms for capping and proofreading. Cell, 2021. 184(13): p. 3474-+.
6. Wang Q., Wu J., Wang H., Gao Y., Liu Q., Mu A., Ji W., Yan L., Zhu Y., Zhu C., Fang X., Yang X., Huang Y., Gao H., Liu F., Ge J., Sun Q., Yang X., Xu W., Liu Z., Yang H., Lou Z., Jiang B., Guddat L.W., Gong P., and Rao Z., Structural Basis for RNA Replication by the SARS-CoV-2 Polymerase. Cell, 2020. 182(2): p. 417-428.e13.
7. Huang Y., Tan L., Liu Y., Zhao H., Wang J., Ge J., Ye S., Liu Z., Lan W., Huang B., Zhang H., Gao Y., Yan L., Rao Z., and Lou Z., Response to: The mechanism for GTP-mediated RNA capping by the SARS-CoV-2 NiRAN domain remains unresolved. Cell, 2025.
8. Wang J., Pan Y., Liu Y., Huang B., Jin G., Zhang L., Zhou F., Chang X., Huang Y., Yan L., Dong Y., Rao Z., Liu D., and Lou Z., Structural Basis and Rational Design of Nucleotide Analogue Inhibitor Evading the SARS-CoV-2 Proofreading Enzyme. J Am Chem Soc, 2025. 147(25): p. 21896-21906.
9. Guo J., Tang X., Huang R., Liu J., and Zhu L., A comprehensive proteomic analysis of umbilical cord blood supports COVID-19 vaccination before pregnancy. Signal Transduct Target Ther, 2024. 9(1): p. 315.
10. Qiu L., Liu X., Xiao M., Xie J., Cao W., Liu Z., Morse A., Xie Y., Li T., and Zhu L., SARS-CoV-2 Is Not Detectable in the Vaginal Fluid of Women With Severe COVID-19 Infection. Clin Infect Dis, 2020. 71(15): p. 813-817.